Deep Pathology
Ziel der Arbeit: Für die medizinische Diagnose ist die Analyse von histologischen Schnitten ein wichtigstes Instrument. Das Anfertigen und Einfärben (engl. staining) der Gewebeproben ist jedoch sehr zeitintensiv und fehleranfällig. In den letzten Jahren sind deshalb virtuelle Einfärbeverfahren in den Fokus der Forschung gerückt, d.h. künstliche neuronale Netze berechnen einen Farblayer für ungefärbte Proben. Diese bestehen meist aus ungelabelten Fluoreszenzbildern, wobei mit der Fluoreszenzlebensdauer-Mikroskopie (FLIM) es neuerdings möglich ist, zusätzliche Informationen über das zeitliche Abklingverhalten der Fluoreszenz zu erhalten. Ziel dieser Arbeit ist es festzustellen, inwieweit FLIM-Daten die Qualität von virtuellen Staining-Algorithmen verbessern können.
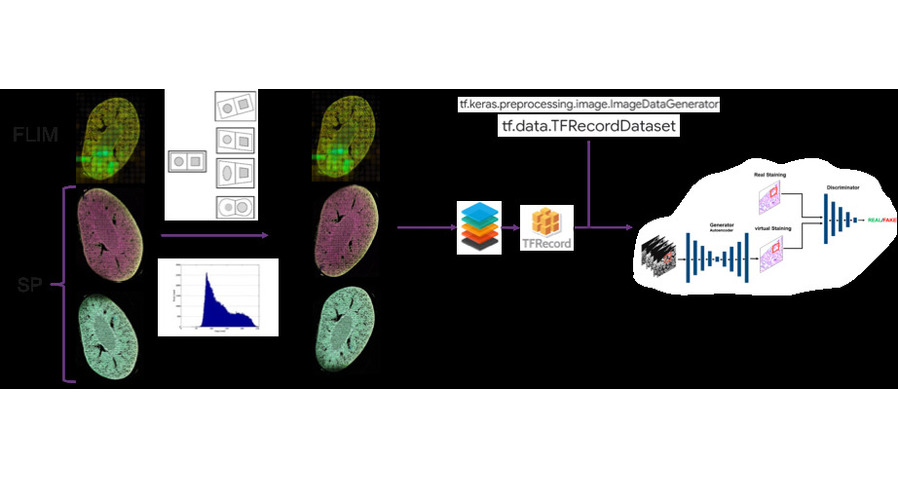
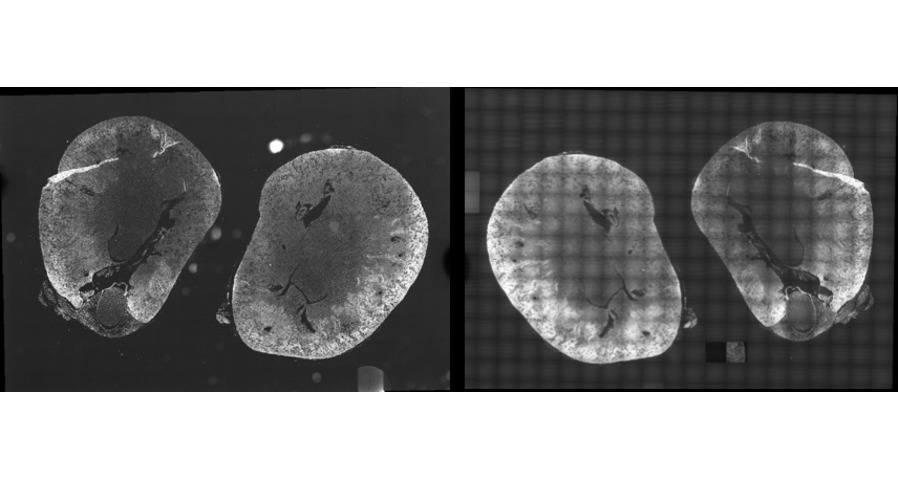
Vorgehen: In dieser Arbeit werden als Grundlage Single-Photon (SP) Fluoreszenzbilder mit verschiedenen Anregefrequenzen und FLIM Bilder als Trainingsinput verwenden. Die SP und FLIM Daten sind unabhängig voneinander aufgenommen worden und besitzen eine unterschiedliche Auflösung. In einem ersten Schritt wurde in dieser Arbeit ein Verfahren zur Registrierung der beiden Modalitäten entwickelt.
In einem zweiten Schritt wurde eine bestehende CycleGAN Netzarchitektur mit dem erweiterten Input-Stack trainiert und Hyperparameter optimiert.
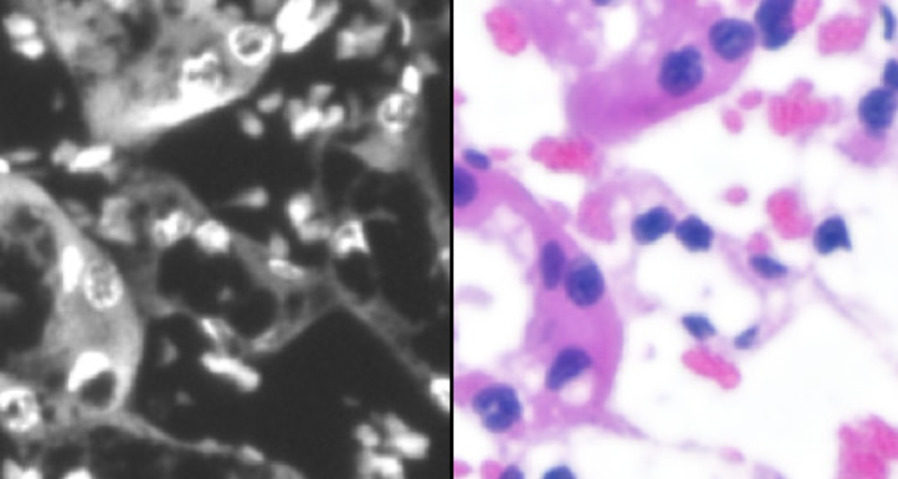
Fazit: Virtuelle Staining-Modelle, die mit den zusätzlichen FLIM Daten trainiert wurden, weisen zusätzliche Farbabstufungen auf im Vergleich zu den Modellen die nur auf SP Daten basieren. Generell wird ein visuell plausibleres Resultat mit dem erweiterten Input erzeugt.